
基因編輯技術(shù)幫助人們實(shí)現(xiàn)了“編輯生命”的愿望��,但該技術(shù)自身存在的局限性也限制了其廣泛應(yīng)用����,此前人們的研究熱情也多集中在編輯的效率、準(zhǔn)確性與安全性��,但常規(guī)的基因編輯技術(shù)在一個(gè)反應(yīng)中僅能對(duì)一個(gè)或數(shù)個(gè)基因進(jìn)行操作�,且通常會(huì)在基因組中留下額外的不需要的序列修改,在大型研究中���,已有的基因編輯技術(shù)遠(yuǎn)遠(yuǎn)無(wú)法滿足大量不同基因的編輯需求�。
為解決上述問(wèn)題�,哈佛大學(xué)George Church教授領(lǐng)導(dǎo)的研究團(tuán)隊(duì)開(kāi)發(fā)了一種基于CRISPR-Cas9的新型高通量基因編輯方法:guide+donor,該方法可以在單個(gè)釀酒酵母中同時(shí)精確改變數(shù)百個(gè)不同基因���,編輯效率達(dá)到80%至100%�,還能在生物體內(nèi)選擇特定行為細(xì)胞�����,進(jìn)行定向基因編輯和修復(fù)。guide+donor除幫助構(gòu)建大型文庫(kù)外����,還能進(jìn)行基因功能分析���,發(fā)現(xiàn)一些未知的基因突變和功能�。
5月21日��,相關(guān)研究成果發(fā)表在著名學(xué)術(shù)期刊Nature Biotechnology上��,文章題為“High-throughput creation and functional profiling of DNA sequence variant libraries using CRISPR–Cas9 in yeast”�。


George Church教授
人類的基因變異通常是點(diǎn)突變導(dǎo)致���,為在不干擾其他潛在變異的情況下重構(gòu)釀酒酵母中的某些變異,CRISPR-Cas9技術(shù)借助sgRNA精確靶向DNA目標(biāo)序列����,在Cas9酶切割其靶序列后,在一個(gè)被稱為同源定向重組(HDR)的過(guò)程中���,借助攜帶目的基因突變的供體模板序列來(lái)修復(fù)基因����。
研究人員表示�,新的研發(fā)策略是將sgRNA、供體模板序列與一個(gè)穩(wěn)定可遺傳的染色體外DNA分子連接起來(lái)�,組成guide+donor組合,這能在一個(gè)反應(yīng)中構(gòu)建大型變體文庫(kù)�,同時(shí)將多個(gè)sgRNAs和供體模板大量傳遞到酵母細(xì)胞,還能定向修復(fù)斷裂的DNA雙鏈�,并通過(guò)NGS技術(shù)識(shí)別被編輯的細(xì)胞。
技術(shù)優(yōu)勢(shì):
在一個(gè)反應(yīng)中快速構(gòu)建大型DNA變異文庫(kù)
在一個(gè)反應(yīng)中同時(shí)提供guide和donor���,防止無(wú)效修復(fù)和非生產(chǎn)性修復(fù)�����。
含有g(shù)uide+donor組合的質(zhì)?���?梢宰鳛樽粉櫥蚓庉嫾?xì)胞的獨(dú)特條形碼,利用NGS技術(shù)可以進(jìn)行高通量分子表型鑒定����。

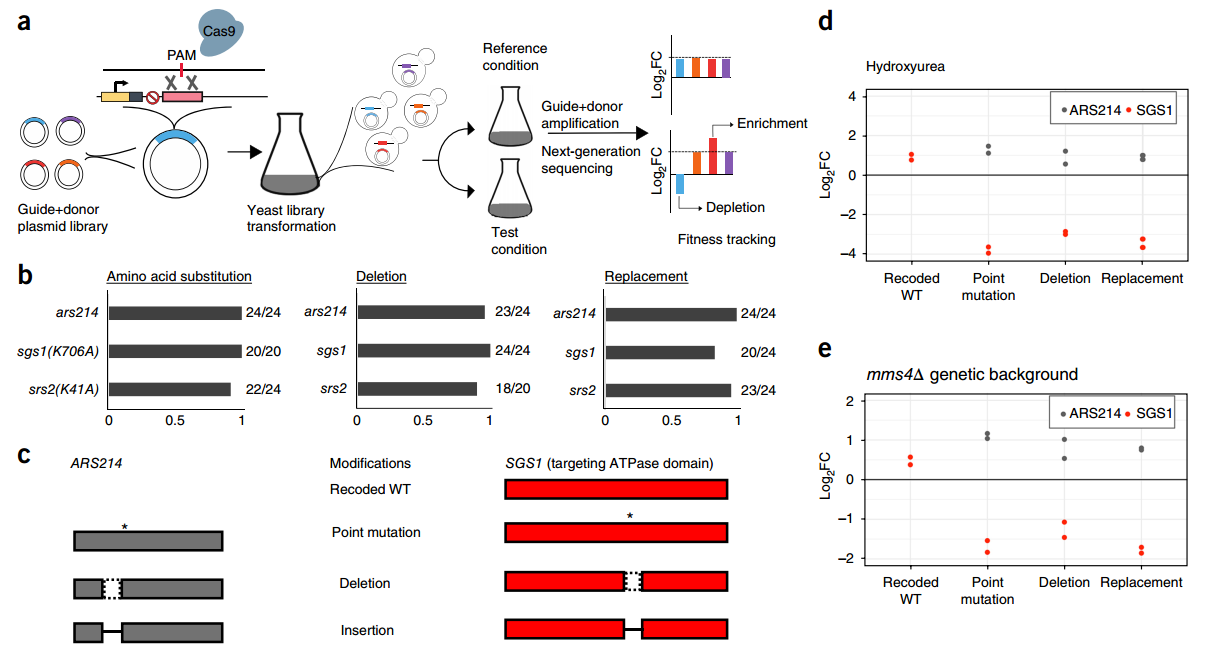
guide+donor基因組編輯平臺(tái)����,來(lái)源:Nature Biotechnology
在證明過(guò)程中�,該團(tuán)隊(duì)首先關(guān)注了酵母中編碼DNA解旋酶和修復(fù)酶SGS1的單個(gè)高保守基因序列,然后他們用一種有毒試劑破壞了攜帶guide+donor文庫(kù)的酵母細(xì)胞DNA����,然后對(duì)存活細(xì)胞進(jìn)行DNA測(cè)序����。最終他們發(fā)現(xiàn)了影響SGS1特征的基因突變�,這些特征對(duì)于受損DNA的修復(fù)至關(guān)重要,并且能影響細(xì)胞持續(xù)存活��。
隨后���,該研究團(tuán)隊(duì)使用guide+donor方法剔除了酵母全基因組中的315個(gè)基因�,它們可以編碼分散在整個(gè)基因組中的小型開(kāi)放閱讀框架(smORF����,長(zhǎng)度<100個(gè)氨基酸)。
在檢測(cè)的315個(gè)smORF中�,研究人員發(fā)現(xiàn)68個(gè)是在細(xì)胞適應(yīng)性存活中起重要作用,這與傳統(tǒng)ORFs功能認(rèn)知相反���,因?yàn)樵谙嗤瑮l件下��,307個(gè)ORF中的104個(gè)會(huì)參與細(xì)胞生長(zhǎng)�����。通過(guò)分析這些基因?qū)湍冈诓煌{迫環(huán)境下存活的影響��,發(fā)現(xiàn)可以將未明確的基本功能分配到特定的smORF��,為基因功能分析打開(kāi)了新的大門(mén)�����。
Church表示��,新的方法不僅能夠更高效�、更準(zhǔn)確地在酵母中進(jìn)行高通量“功能基因組學(xué)”研究,還能模擬���、檢測(cè)酵母細(xì)胞中與特定性狀或功能紊亂相關(guān)的低頻基因突變,并找出哪些與疾病實(shí)際相關(guān)���。此外�����,該方法除了可以在龐大的基因家族中挖掘新的基因和功能外���,還能研究基因組中的非編碼序列�����,提高我們對(duì)基因調(diào)控和染色體生物學(xué)的理解�。
參考文獻(xiàn):
1.Profiling the genome hundreds of variations at a time
2.High-throughput creation and functional profiling of DNA sequence variant libraries using CRISPR–Cas9 in yeast
轉(zhuǎn)自:測(cè)序中國(guó)